http://dx.doi.org/10.18667/cienciaypoderaereo.531
Artículo
CIENCIA Y PODER AÉREO 11(1): 194-201, 2016
Software "Biomepro" para acondicionamiento de señales EMG superficiales Y obtención de parámetros relevantes1
Software "Biomepro" para acondicionamento de sinais EMG superficiais e obtenção de parámetros relevantes2
"Biomepro" Software For The Conditioning Of Superficial EMG Signals And Obteneion Of Relevante Parameters3
Robin Blanco,4 Andrés Cifuentes,5 Mauricio Plaza6
Universidad Militar Nueva Granada
Recibido: 21/08/2016
Aprobado evaluador interno: 02/09/2016
Aprobado evaluador externo: 12/09/2016
Para citar este artículo: Blanco, R., Clfuentes, A.., & Plaza, M. (2016). Software BIOMEPRO para acondicionamiento de señales EMG superficiales y obtención de parámetros relevantes. Ciencia y Poder Aéreo, 11 (1), 194-201. Doi: http://dx.doi.org/10.18667/cienciaypoderaereo.531
Resumo: no presente artigo divulga se o desenvolvimento de BloMePro, uma ferramenta de software para o processamento de a mostras de eletromlografia (EMG) superficial, implementado em JAVA, uma linguagem multi-plataforma orientada para objetos e de código interpretado, linguagem escolhida para facilitar a portabilidade do software, mencionado. BioMePro facilita o trabalho de extração de características de sinais EMG superficiais, úteis para sistemas de Inferência quanto redes neuronals ou maquinas de suporte vetorial, a partir duma interfase clara e fácil de usar com um conjunto variado de opções que permitem eleger o melhor processamento que é requerido para uma aplicação especifica. Como sistema de aquisição dos dados se usou o equipamento BiosignalPlux®, os sinais de EMG de prova do software que foram capturados por melo da implementação dum protocolo de registro de movimentos de pronação no antebraço direito em pacientes jovens e sadios, protocolo elegido pela sua simplicidade de Implementação e confiabilidade nas a mostras.
Palavras-chave: eletromlografia; envoltura RMS; Java; processamento de sinais.Abstract: This article presents the development of BioMePro, a software tool for the Processing of superficial electromyography (EMG) samples, implemented in JAVA, a multiplatform language, object oriented and interpreted code, language chosen in orderto facilitate the portability of the Software in mention. BioMePro facilitates the work of extracting characteristics of superficial EMG signáis, useful for inference systems such as neural networks or vector support machines, from a clear and easy to use interface with a varied set of options that allow to select the best Processing that is requlred for a specific application. As a data acquisition system, the BiosignalPlux® equipment was used,the EMG test signáis from the software that werecaptured through the implementation of a protocol for recording right forearm pronation movements in healthy young patlents, a protocol chosen by its simplicity of implementation and reliability in the samples.
Key Words: Electromyography; Java; RMSWrappIng; Signal Processing.Introducción
El desarrollo de herramientas de software para el procesamiento de bioseñales ha venido en aumento, debido a las ventajas que ha mostrado para el apoyo del personal médico en el diagnóstico, tratamiento y paliación de cualquier afectación a la salud de las personas (Kazerooni & Strausser, 2011). Dichos programas van desde aquellos que solamente graban y/o visualizan las bioseñales, hasta programas que procesan los datos y obtienen parámetros importantes para un diagnóstico; como, por ejemplo, la frecuencia cardíaca y la frecuencia respiratoria.
Estas herramientas son muy útiles para el desarrollo de investigaciones en el área médica y su posterior aplicación práctica (Romo, Realpe, & Jojoa, 2007) (Figueroa-García, y otros, 2014) (Momen, Krishnan, & Chau, 2007). En etapas de investigación tempranas suele no ser favorable la adquisición de una amplia gama de estas herramientas, por el costo de algunas, que se suma al de los equipos de adquisición de bioseñales (Baturone & Gersnoviez, 2010). La idea de herramientas de software portables, de bajo costo, para el procesamiento complejo de bioseñales es una idea llamativa para una gran cantidad de proyectos que requieran la obtención de información de señales electrofisi-ológicas, para aplicaciones más especializadas.
Una de las bioseñales más procesadas en el ámbito de la investigación biomédica, es la de electromiografía (EMG) (Figueroa-García, y otros, 2014) (Saikia, Mazumdar, Sahai, Paul, & Bhatia, 2016), la cual corresponde a la medición de la actividad eléctrica en el músculo debida a una acción de movimiento, la información obtenida de esta clase de señales ha sido muy útil en desarrollos ingeníenles como interfaces para el control de sistemas robóticos tipo ex-oesqueletos (Kazerooni & Strausser, 2011). Esta señal se puede medir de forma superficial sin generar mayor incomodidad al paciente en estudio, ni comprometer la riqueza de la información relacionada con la medición. A la hora de trabajar con señales de EMG superficiales es necesario considerar el hecho de que en esta no solo se encuentra la información de la actividad eléctrica de una única célula muscular o unidad motora, sino que se registra la suma de muchas actividades de las células cercanas a la colocación del electrodo, por lo tanto, al visualizar la señal, esta suele presentar un patrón similar al ruido eléctrico, lo que obliga a definir una estrategia para obtener parámetros relevantes que indiquen información clara del movimiento en estudio (Saikia, Mazumdar, Sahai, Paul, & Bhatia, 2016).
Metodología
El desarrollo de la herramienta de software BioMePro (Universidad Militar Nueva Granada, 2016) partió de unos requerimientos iniciales obtenidos de la interacción directa con investigadores del área para conocer las necesidades relacionadas con adquisición y procesamiento de bioseñales en sus respectivos proyectos; estos requerimientos suelen conocerse como requerimientos de cliente. En este caso para el software diseñado las necesidades encontradas fueron:
Estos requerimientos fueron objetivos a cumplir en el diseño, para lo cual se realizó un seguimiento en las pruebas finales del software con el fin de corroborar si fueron satisfechos y evaluar la calidad de ello.
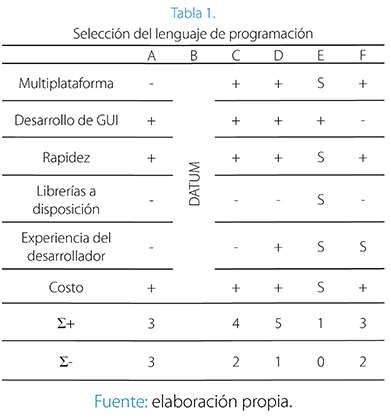
En el proceso de diseño, para la selección del lenguaje de programación, la metodología de selección empleada fue la matriz PUGH (Dieter, 2009), los elementos enfrentados para comparación fueron diferentes lenguajes de programación comunes en el desarrollo de esto tipo de aplicaciones, la aplicación del método se resume en la siguiente tabla:
Donde las opciones de lenguajes de programación que se evaluaron fueron:
Se observó que la opción predominante para los criterios de diseño elegidos fue JAVA. En una discusión al interior del grupo de diseño, este lenguaje fue seleccionado sin aplicar metodologías de selección adicionales por su característica de multiplataforma, que permite al desarrollo explicado en el presente artículo su extrapolación sencilla a proyectos en diferentes plataformas de software.
En la etapa de materialización del diseño de software se definieron los datos de entrada, los cuales correspondieron a registros de señales de EMG superficiales del músculo pronador redondo en antebrazo derecho, realizando movimientos de pronaclón siguiendo el protocolo propuesto en (Robín, Andrés, & Mauricio, 2016). La adquisición de la muestra para las pruebas de funcionamiento se realizó por medio de electrodos superficiales de AgCI en configuración bipolar (Hargrove, Englehart, & Hudgins, 2007), en este caso 3 electrodos fueron empleados, 2 sobre la parte media del músculo y uno sobre la cabeza distal del hueso cúbito Figura 1.
Se utilizó la herramienta BiosignalPlux® para la adquisición de señales de EMG superficiales, la cual se configuró a una frecuencia de muestreo de 1 KHz y a una resolución de 16 bits, los datos obtenidos son enviados a un PC en donde el software Opensignals®, de descarga libre, guarda los datos en archivos con extensión TXT para ser abiertos luego por el software BioMePro. Este desarrollo fue hecho en el entorno NetBeans®, en donde se realizaron la escritura, compilación y depuración del programa.
Las fundones principales del software diseñado se listan a continuación:
Para lograr los requerimientos del software propuestos, se diseñó una interfaz gráfica consistente de 10 clases (Figura 2). La clase principal configura los parámetros iniciales de funcionamiento del software, la clase correspondiente a la interfaz gráfica principal (diferente a la clase principal) contiene todos los componentes gráficos y eventos con los que interactúa el usuario, 7 clases adicionales de tipo interfaz gráfica, consistentes en cuadros de diálogo contienen la implementación de los módulos de funcionamiento propios del software, y finalmente, una clase adicional, contiene y comparte los datos con las demás clases por medio de la asignación de referencias a objetos. La interfaz gráfica de usuario se presenta en la Figura 3.
Los 7 diálogos presentados en la Figura 2, son los módulos internos del software los cuales comparten la misma referencia a la clase "datos" donde se encuentran los datos sin procesar y los datos del proceso de filtrado y armonizado. Una descripción breve de los diálogos se presenta a continuación:
Diálogo abrir: aparte de importar los datos, realiza la conversión a miliVoltios del valor entregado por el openSignals®, debido a que los datos registrados por el BiosignalPlux® vienen en unidades enteras del ADC del equipo. Esta conversión se logra con un ajuste lineal a partir de 2 parámetros: span (pendiente) y offset (desplazamiento), que son preguntados al usuario.
Diálogo guardar: permite construir un archivo de texto plano con los parámetros extraídos de la señal y su habilitación por parte del usuario.
Diálogo Filtro PasaBanda: dependiendo la frecuencia de muestreo ingresada por el usuario en la interfaz gráfica, este didlogo le indica la respuesta en frecuencia del filtro pasabanda pre programado.
Diálogo filtro notch: dependiendo de la frecuencia de muestreo, este didlogo le indica la respuesta en frecuencia del filtro, rechaza banda tipo Notch predeterminado y espera su confirmación para ser aplicado.
Diálogo envoltura D.C.: este didlogo realiza un armonizado de la señal, consistente en reemplazar cada valor en la muestra por el promedio de la suma de los valores absolutos de los datos en su vecindad, el tamaño de la vecindad debe definirse menor al tamaño total de la muestra y suele ser un tamaño significativamente menor (Universidad Militar Nueva Granada, 2016).
Diálogo envoltura RMS: este didlogo permite un armonizado de la señal a partir del cálculo del valor cuadrática medio de una pequeña ventana, de forma similar a lo explicado en el ítem anterior. (Universidad Militar Nueva Granada, 2016).
Diálogo FFT: realiza la Transformada rápida de Fourier (del inglés Fast Fourier Transform FFT), usando librerías y códigos para Java, desarrollados en Princeton University (Princeton University, 2016). Se debe tener cuidado de que el tamaño de la muestra a analizar corresponda a una potencia de 2, los resultados son presentados en un diagrama de magnitud de la FFT.
El banco de filtros fue diseñado en Matlab®, y está compuesto de 8 pares de filtros: un pasabanda y una notch. Son 8 pares porque hay 8 diferentes frecuencias de muestreo que el software admite: 300, 400, 500, 600, 700, 800, 900 y 1000 Hz. Los filtros pasabanda son de tipo FIR {Finite Impulse Response), diseñados para cada frecuencia de muestreo usando el método de filtros óptimos de rizado constante (Dipen, Shashwat, Siddharth, Sukrut, & Bhavdip, 2015), con una banda de paso de 50 a 150 Hz aproximadamente, ancho seleccionado debido a que en este rango se encuentra la información relevante para EMG (Robín, Andrés, & Mauricio, 2016). Los filtros notch son de tipo MR (Infinite Impulse Response) y fueron diseñados para rechazar la frecuencia de 60Hz presente en el sistema eléctrico de Colombia. Como estos filtros ya estaban preestablecidos al usuario, solo se le preguntó si los deseaba aplicar al mostrarle su respuesta en frecuencia por medio del diagrama de Bode de amplitud Figura 4.
Análisis de resultados
Se observaron las gráficas de la muestra importada por el software, con sus respectivas opciones de acondicionamiento, como se muestra en la Figura 5, estos resultados gráficos fueron comparados con las gráficas obtenidas en Matlab® Figura 6.
Los parámetros relevantes obtenidos (Valor DC, Valor RMS,Tiempo de Duración y Frecuencia Pico) se guardaron en archivos de texto plano (Figura 7), para su uso posterior en otro tipo de programa que requiera los parámetros que caracteriza a los registros de señales de EMG.
También se compararon los tiempos de ejecución entre el software BioMePro y el programa ya realizado en Matlab®, realizando las operaciones de filtro pasabanda y notch, y las armonizaciones por envoltura DC y RMS. Se obtuvieron los datos de la siguiente tabla:
Se observa que, aunque el proceso del filtrado pasa-banda es más demorado en el software BloMePro, sin embargo, en el total de las operaciones el ahorro de tiempo es considerable y más si se proyecta implementar a futuro en aplicaciones en tiempo real.
Conclusiones
Se obtuvo un programa de características similares a códigos realizados en Matlab® con la ventaja de que ahora son multlplataforma y son de registro libre porque ya no se requiere de la compra de la licencia del lenguaje de programación. Además, se logró un ahorro de tiempo considerable, el cual se puede aprovechar más si se consideran propiedades de simetría en los coeficientes del filtro FIR para la operación de filtrado pasabanda.
Un aspecto Importante que se ganó es que a la hora de comparar el peso del código realizado en Matlab® donde se obtuvo 44KB comparado al ejecutable BioMePro de 2,8 MB no parece ventajoso, pero si es analizado que ya no se requiere el software Matlab® para el procesamiento de las señales EMG, el cual es muy pesado e Impensable colocar en un sistema embebido, el programa permite ganar la característica de portabllldad y de implementaclón, por ejemplo, en un RaspBerry Pi®.
Se comprobó que el programa corrió en varias plataformas Windows y Linux sin necesidad de Instalar las librerías usadas en cada computador, comprobando la característica de multlplataforma de Java que era un requerimiento planteado para el software.
Este software puede ser Implementado en trabajos posteriores como el diseño de señales de control para sistemas robótlcos tipo exoesqueletos, que requieren de un procesamiento muy robusto de señales electrofisiológi-cas como las EMG para la Interacción con el usuario, que requieran posibilidades de procesamiento embebido.
1 Artículo científico original derivado del proyecto IMP-ING-2135 titulado "Diseño de dispositivo exoesqueléti-co mecánico para ayuda de personas con problema de tensión muscular y actividades repetitivas de la Universidad Militar Nueva Granada (UMNG), Bogotá D.C., Colombia (2016).
2 Artigo científico original derivado do projeto IMP-ING-2í35 com título "Desenho de um dispositivo exoesquelético mecánico para ajuda de pessoas com problemas de tensão muscular e atividades repetitivas da Universidade Militar Nueva Granada (UMNG), Bogotá D.C., Colombia (2016).
3 Original scien tifie article derived from the IMP-ING-2135 project titled "Design of mechanical exoes-keletal device to help people with muscular tensión problems and repetitive activities ofthe Nueva Granada Military University (UMNG), Bogotá D.C., Colombia (2016).
4Ingeniero Mecatrónlco de la Universidad Nacional de Colombia, Estudiante de la Maestría en Ingeniería Mecatrónlca de la Universidad Militar Nueva Granada. Correo electrónico: u3900208@unimilitar.edu.co
5 Ingeniero Mecatrónlco de la Universidad Nacional de Colombia, Magíster en Ingeniería Mecatrónlca de la Universidad Militar Nueva Granada. Correo electrónico: andres.cifuentes@unimilitar.edu.co
6 Ingeniero Eléctrico Universidad de los Andes, Doctor en Ingeniería con énfasis en Realidad Virtual y Blolngenlería. Correo electrónico: mauricio.plaza@unimilitar.edu.co
Referencias
Baturone, I., & Gersnovlez, A. (2010). Diseño de Sistemas Neu-ro-DIfusos para Control de Robots mediante XFUZZY 3. Tecnología, aprendizaje y enseñana déla electrónica. Sevilla.
Blanco, R. (2016). Diseño de un sistema de predicción del rendimiento deportivo a partir de técnicas de inteligencia computacional en selecciones UMNG. Bogotá: Propuesta de Tesis, Maestría en Ingeniería Mecatrónlca, Universidad Militar Nueva Granada.
D. K., S. G., S. K., S. G., & B. G. (2015). Deslgn of Equlrlpple Lln-ear-Phase FIR Fllter Uslng Computer Alded Method. Communications and SignaI Processing (tCCSP), (págs. 0001 -0005). Melmaruvathur, India.
DletepG. E. (2009). Engineering Design. Boston: McGraw-FIIII.
Flgueroa-Garda, I., Aguilar-Leal, O., Hernández-Reynoso, A., Madrigal, J., Fuentes, R., Huegel, J., & García-Gonzalez, A. (2014). Platform for the Study of Virtual Task-Oriented Motion and its evaluation by EEG and EMG Biopotentials. IEEE Engineering in Medicine and Biology Society, 36th Annual International Confer-ence. Chicago, USA.
Hargrove, L.Englehart, K., & Hudgins, B. (2007). A Comparison of Surface and Intramuscular Myoelectrlc Signal Classlficatlon. IEEE transactions on biomedical engineering, vol. 54, no. 5.
Kazeroonl, H.,&Strausser, K. A. (2011). The DevelopmentandTest-ing of a Human Machine Interface fora Mobile Medical Exo-skeleton. 2011IEEE/RSJInternational Conference on Intelligent Robots and Systems. San Francisco, USA.
Momen, K., Krlshnan, S., & Chau, T. (2007). Real-Time Classlficatlon of Forearm Electromyographlc Signáis Correspondlng to User-Selected Intentlonal Movements for Multlfunctlon Prosthesls Control. IEEE Transactions on Neural Systems and Rehabilitation Engineering, 15(4), 535-542.
Princeton University. (16 de 11 de 2016). Introduction ToProgram-ming in Java. Obtenido de http://introcs.cs.princeton.edu/ java/97data/
R. B., A. C, & M. P. (2016). Diseño de un sistema difuso para el reconocimiento de la actividad muscular. IJIAS, 19(2).
Romo, H. A., Realpe, J. C, & Jojoa, P. E. (2007). Análisis de Señales EMG Superficiales y su Aplicación en Control de Prótesis de Mano. 14(1).
Saikia, A., Mazumdar, S., Sahai, N., Paul, S., & Bhatla, D. (2016). Com-paratlve Study and Feature Extractlon of the Muscle Actlvlty Patterns in Healthy Subjects. 3rd International Conference on Signa! Processing and integrated Networks. Nolda, India.
Universidad Militar Nueva Granada. (2016). Manual de Usuario de Software BioMePro. Bogotá.